Descifrando las funciones de los microbiomas en Ingeniería Ambiental
Introducción
Un microbioma es la comunidad de microorganismos (bacterias, arqueas, hongos, virus, etc.) que habita en un entorno específico. En las últimas décadas, el estudio de los microbiomas ha avanzado significativamente. Si bien los microbiomas humanos, como el intestinal y el cutáneo, han recibido gran atención, los microbiomas ambientales —presentes en suelos, aguas y aire— son igualmente importantes por su papel en los ciclos biogeoquímicos.
En ingeniería ambiental, estos microbiomas, también conocidos como comunidades microbianas, consorcios microbianos o cultivos mixtos, son fundamentales para la optimización de bioprocesos. Estos bioprocesos son utilizados en el tratamiento de aguas residuales, la biorremediación, la producción de biocombustibles y la valorización de residuos. Comprender el rol de los microorganismos en un microbioma dentro de un bioproceso es clave para mejorar la eficiencia de estos procesos desde la ingeniería.
Herramientas de Secuenciación
En los últimos años, las herramientas de secuenciación de alto rendimiento han transformado nuestra comprensión de la ecología microbiana, incluyendo la diversidad, distribución y abundancia de microorganismos en distintos entornos, así como sus interacciones. Sin embargo, aún existe un gran desafío en relacionar microorganismos específicos con sus funciones dentro del ecosistema.
Existen varias técnicas de secuenciación. La más completa es la secuenciación shotgun, que permite analizar todo el ADN presente en una muestra de microbioma. A diferencia de métodos que se enfocan en regiones específicas, esta técnica fragmenta el ADN en piezas pequeñas que luego se secuencian y ensamblan para reconstruir los genomas completos de los microorganismos presentes, generando datos metagenómicos.
No obstante, esta técnica requiere una inversión considerable en tiempo y recursos económicos, además de herramientas bioinformáticas avanzadas para procesar y analizar los datos (Djemiel
et al., 2022).
Otra técnica de secuenciación es el metabarcoding, que utiliza regiones específicas del ADN, como el gen 16S rRNA en bacterias, para identificar las especies presentes. A diferencia del enfoque shotgun, que secuencia todo el ADN de la muestra, el metabarcoding se concentra en segmentos que actúan como "códigos de barras" para diferentes especies. Esta técnica es más rápida y económica, lo que facilita estudios a gran escala con recursos más limitados (Djemiel
et al., 2022).
Predicción Funcional
La predicción de las funciones de un microbioma se puede realizar con datos provenientes de secuenciación shotgun o de metabarcoding basado en el gen 16S rRNA. A continuación, se describen brevemente los métodos empleados para cada tipo de datos:
Datos metagenómicos:
Los datos generados por secuenciación shotgun se comparan con bases de datos genómicas que contienen secuencias de genomas completos de organismos conocidos, permitiendo la identificación de los microorganismos presentes en la muestra. Luego, estos datos se analizan con bases de datos funcionales, lo que permite traducir la información genética en funciones específicas, como la degradación de compuestos orgánicos o la fijación de nitrógeno. Existen herramientas especializadas para este propósito, como HUMAnN (HMP Unified Metabolic Analysis Network; (Abubucker
et al., 2012)), que utiliza bases de datos genómicas como UniRef y bases de datos funcionales como MetaCyc.
Datos de secuenciación 16S rRNA:
En este caso, las secuencias obtenidas se comparan con bases de datos de referencia, como SILVA o Greengenes, para la identificación taxonómica de los microorganismos. Herramientas como PICRUSt (Phylogenetic Investigation of Communities by Reconstruction of Unobserved States; (Douglas
et al., 2020)) y Tax4Fun permiten inferir funciones microbianas a partir de estas identificaciones, usando bases de datos funcionales como KEGG y MetaCyc para mapear las secuencias a genes y funciones conocidas. Este enfoque es más económico, aunque ofrece una menor resolución en comparación con los datos metagenómicos.
Para entender mejor la predicción funcional, es importante conocer cómo se conectan los genes con las funciones observadas en un microbioma (Fig. 1):
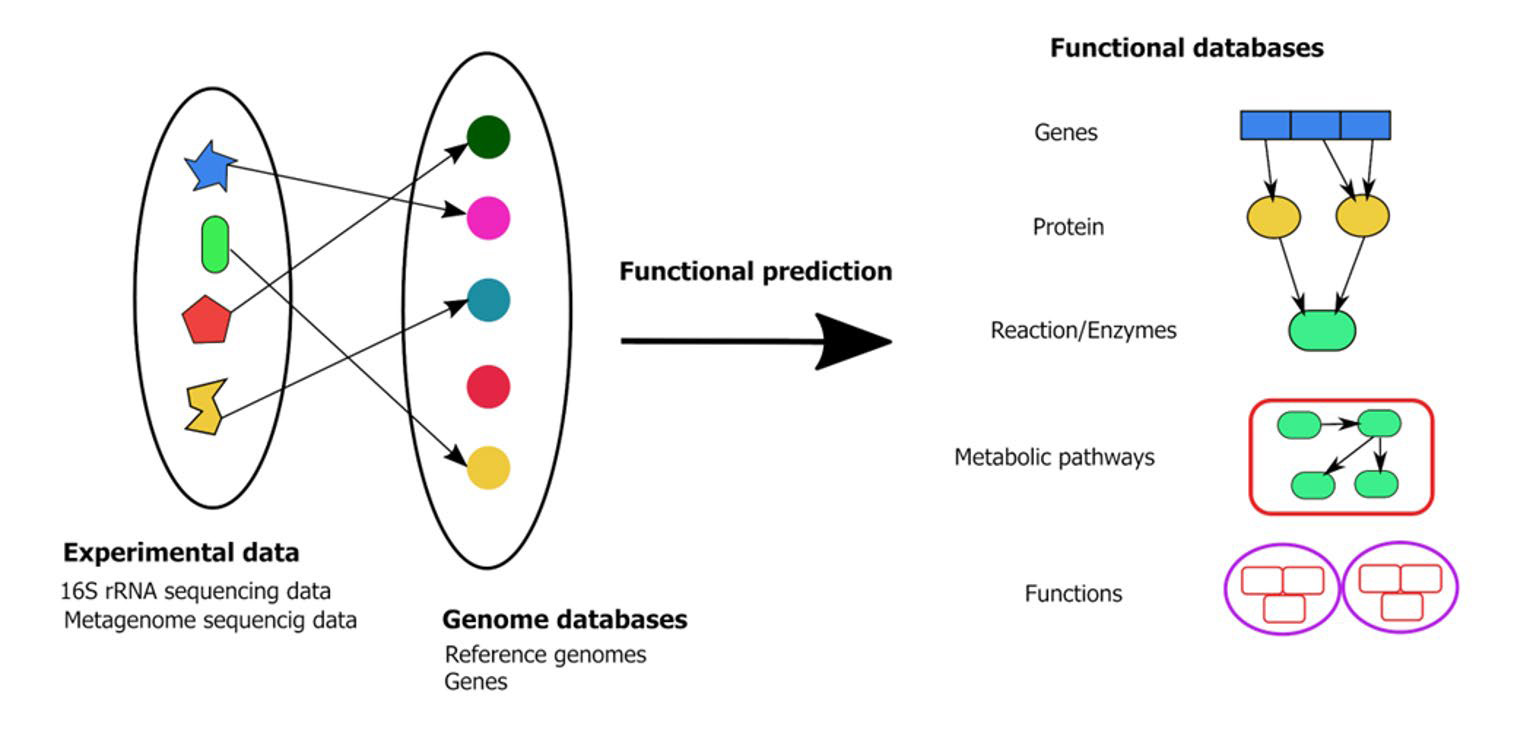
Fig. 1. Predicción funcional a partir de datos experimentales
provenientes de la secuenciación del ADN.
• Genes: Segmentos de ADN que contienen instrucciones para construir proteínas.
• Proteínas: Moléculas formadas por aminoácidos que desempeñan funciones cruciales en los organismos.
• Enzimas: Proteínas que actúan como catalizadores en reacciones bioquímicas.
• Reacciones: Procesos bioquímicos dentro de las células facilitados por enzimas.
• Vías Metabólicas: Series de reacciones bioquímicas interconectadas que transforman moléculas.
• Funciones: Actividades metabólicas colectivas que surgen de la interacción entre diferentes vías metabólicas presentes en microorganismos.
Nuevas herramientas en el Instituto de Ingeniería
La Unidad Académica Juriquilla participa en el proyecto “Cambio de paradigma: residuos como materia prima para conciliar el eje agua-energía-ambiente-seguridad alimentaria" (Grupos Interdisciplinarios de Investigación del Instituto de Ingeniería, UNAM, No. 3406). Este proyecto busca proponer y evaluar procesos para la valorización de residuos, alineados con la economía circular. La meta es convertir residuos orgánicos en materia prima para producir energía renovable, productos de valor agregado y compuestos químicos que puedan ser utilizados en diversos sectores.
Dentro de este proyecto, el paquete de trabajo I3, enfocado en la microbiología de procesos, aplica herramientas genómicas y metagenómicas para optimizar procesos anaerobios de valorización de residuos. En este contexto, los que participamos en este proyecto, hemos incorporado a nuestra cadena de procesos para el análisis de comunidades, el análisis de perfiles funcionales. Este análisis se realiza mediante la herramienta PICRUSt2 lo que permite predecir funciones metabólicas a partir de secuenciación 16S rRNA. Actualmente, también estamos en la tarea de integrar herramientas para el análisis funcional a partir de datos metagenómicos.
El uso de estas herramientas ha contribuido al desarrollo de nuevo conocimiento científico de alta calidad. Por ejemplo, hemos investigado procesos como la producción de metano a partir de efluentes de la industria vinícola y la generación de hidrógeno a partir de residuos lácteos (Muñoz-Páez et al., 2024; Vital-Jácome et al., 2024). El conocimiento generado sobre las funciones de los microorganismos en estos procesos complementa otros estudios de ecología microbiana y permite formular nuevas hipótesis sobre las interacciones en los microbiomas. La Fig. 2 y Fig. 3 muestran ejemplos del tipo de resultados obtenidos mediante el análisis funcional en dos casos de estudio.
Conclusión y perspectivas
El estudio de los microbiomas en ingeniería ambiental está abriendo nuevas posibilidades para el desarrollo de bioprocesos más eficientes y sostenibles. A medida que las técnicas de secuenciación, como la: secuenciación shotgun y el metabarcoding, y las herramientas bioinformáticas continúan avanzando, podemos obtener un conocimiento más detallado sobre la relación entre la diversidad microbiana y sus funciones. Este conocimiento ya se está utilizando para complementar investigaciones en otras disciplinas con enfoques integradores, como la biología de sistemas y la ingeniería metabólica, que en el futuro podrían permitir el diseño de microbiomas a medida para aplicaciones específicas en ingeniería ambiental. La integración de estos enfoques con nuevas tecnologías de ingeniería podría revolucionar la forma en que gestionamos los recursos naturales y abordamos los desafíos ambientales, contribuyendo significativamente a la sostenibilidad global.
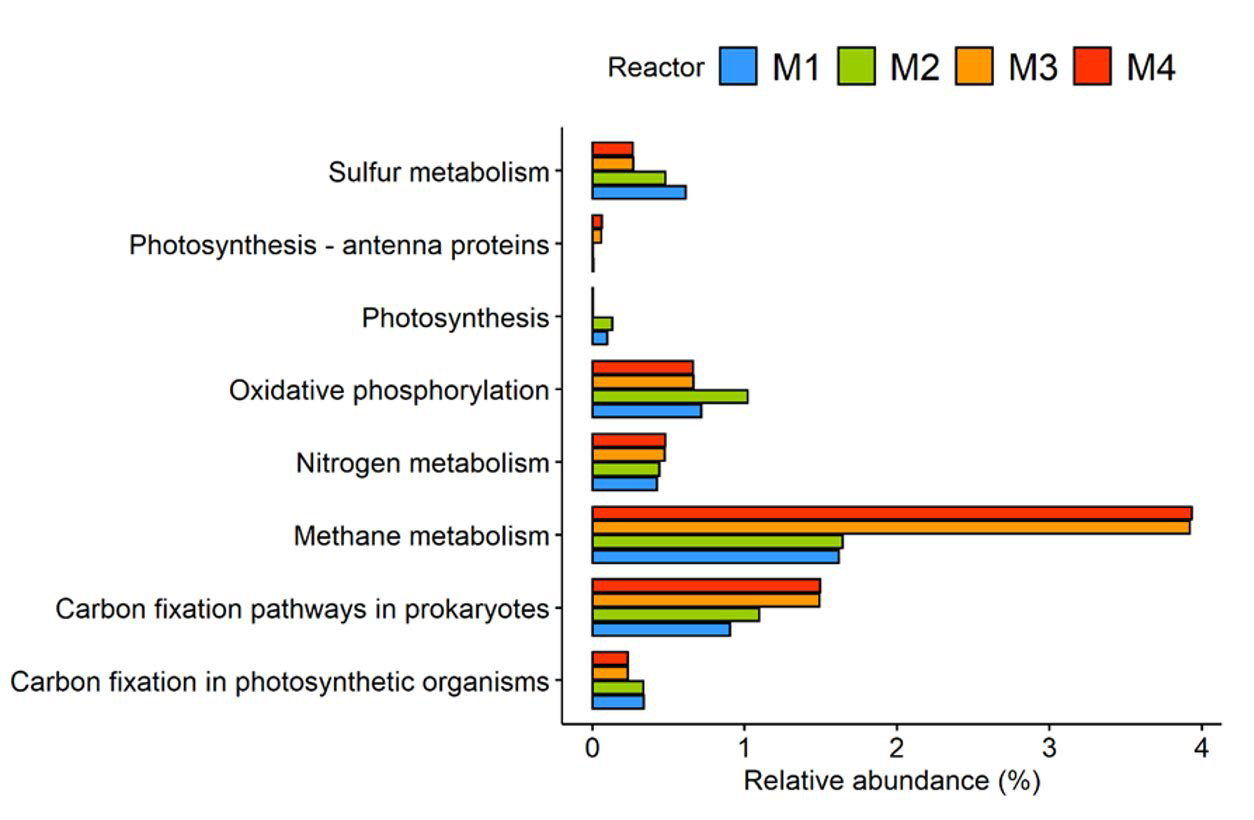
Fig. 2. Abundancia relativa de diferentes tipos de metabolismo, predicha para 4 configuraciones de reactor productor
de metano a partir de efluentes de la industria del vino (M1-M4). Fuente: Vital-Jácome et al., 2024.
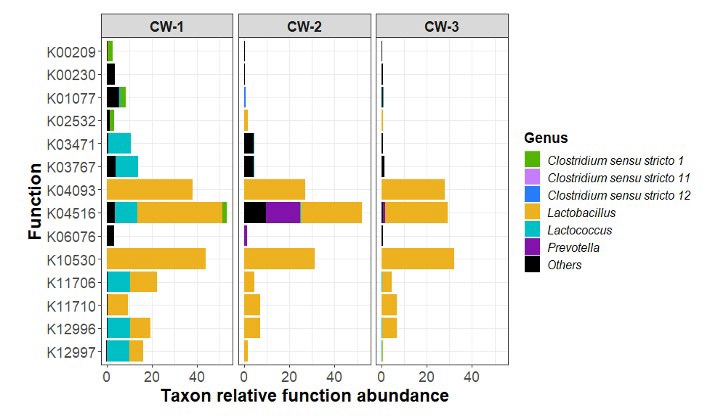
Fig. 3. Abundancia relativa por género de microorganismo, predicha específica para diferentes enzimas (Function) y para 3 condiciones
de operación de un mismo reactor productor de hidrógeno a partir de suero de queso (CW-1-3). Fuente: Muñoz-Páez et al., 2024.
Referencias
Abubucker, S., Segata, N., Goll, J., Schubert, A.M., Izard, J., Cantarel, B.L., Rodriguez-Mueller, B., Zucker, J., Thiagarajan, M., Henrissat, B., White, O., Kelley, S.T., Methé, B., Schloss, P.D., Gevers, D., Mitreva, M., Huttenhower, C.,2012. Metabolic Reconstruction for Metagenomic Data and Its Application to the Human Microbiome. PLOS Comput. Biol. 8, e1002358.
https://doi.org/10.1371/JOURNAL. PCBI.1002358
Djemiel, C., Maron, P.A., Terrat, S., Dequiedt, S., Cottin, A., Ranjard, L., 2022. Inferring microbiota functions from taxonomic genes: a review. Gigascience 11, 1–30.
https://doi.org/10.1093/gigascience/giab090
Douglas, G.M., Maffei, V.J., Zaneveld, J.R., Yurgel, S.N., Brown, J.R., Taylor, C.M., Huttenhower, C., Langille, M.G.I., 2020. PICRUSt2 for prediction of metagenome functions. Nat. Biotechnol. 38, 685–688.
https://doi. org/10.1038/s41587-020-0548-6 Muñoz-Páez, K.M., Buitrón, G., Vital-Jácome, M., 2024. Predicting metabolic pathways and microbial interactions in dark fermentation systems treating real cheese whey effluents. Bioresour. Technol. 413. https://doi.org/10.1016/j.biortech.2024.131536
Vital-Jácome, M., Carrillo-Reyes, J., Buitrón, G., 2024. Metabolic Functional Profiles of Microbial Communities in Methane Production Systems Treating Winery Wastewater. Bioenergy Res. 17, 669–680.
https://doi. org/10.1007/s12155-023-10633-3